|
|
 |
| јвторизаци€ |
|
|
 |
| ѕоиск по указател€м |
|
 |
|
 |
|
|
 |
 |
|
 |
|
| Voit E. Ч Computational Analysis of Biochemical Systems: A Practical Guide for Biochemists and Molecular Biologists |
|
|
 |
| ѕредметный указатель |
Rapoport, S.M. 6 40 61 119 251 474 476
Rapoport, T.A. 6 60 61 119 245 251 476
Rapp, B.A. 18 470
Rat liver 148 153 297 327
Rat liver, glycolytic-glycogenolytic pathway in 365Ч398
Rat liver, pyruvate carboxylate in 151Ч152
Rat liver, pyruvate kinase in 150Ч151
Rate constants 44Ч46 59 65Ч67 76 79 81 88 90 94 95 101 103 105 110 115 120 126 127 130 134 142 143 146 147 176 177 179 183 188 191 192 194 196Ч198 200 201 205Ч207 214 217 219 220 222 227 230 233 242 244Ч247 249 252 265 268 269 271Ч274 278 287 291 298 299 303 305 308 311Ч314 318 320 324 328 338Ч340 342 350 351 353Ч355 358 363 368 370 377 379 380 396Ч398 445 446 448 449 452 455Ч457 461Ч463 466 467
Rate constants in fluxes 78Ч79 82 254Ч255
Rate constants in GMA models 77Ч78 254Ч255
Rate constants in metabolite concentrations 235Ч241 254
Rate constants, branch point constraints 393Ч395
Rate constants, constrained systems 239Ч241 391Ч395
Rate constants, degradation 50 123 267
Rate constants, estimation of 164Ч171
Rate constants, first-order 308
Rate constants, glucose-6-phosphate 122Ч123 167Ч171
Rate constants, in glycolytic-glycogenolytic pathway model 375 390Ч395
Rate constants, in S-systems 50 51Ч52 53 70 77 78
Rate constants, indexed 77
Rate constants, isocitrate in Dictyostelium discoideum 165Ч167
Rate constants, precursor-product relationships 391Ч393
Rate constants, sensitivities with respect to 122Ч123 235Ч241 254Ч255 375 390Ч395
Rate constants, symbols 50
Rate constants, unconstrained parameters 391
Rate laws 2 6 40 47 50 58 67 142 144Ч146 150 159 160Ч164 167 168 187 189 246 248 263 270 273 274 276 295Ч297 305 330 350 361 362 406 418 419
Rate laws in enzyme-catalyzed reactions 53
Rate laws, Michaelis Ч Menten 37 38 42Ч43 74 83
Rate laws, number of terms 39
Rate laws, power functions and 55 74
Rate laws, representations of 16
Rate laws, traditional 37 41 60 92 165 169 186 188 260 265 268 269 272 275 277 283 291 304 338 355
Rational functions 38 50
Reactions activation 14 27 28 86 112Ч113
Reactions occurring on surfaces 53
Real part 124 125 126 210 212 213 216 280 307 340 376 379 448
Realistic conditions 40
Recasting 10 137 142 409Ч410 411
Recker, D. 359 473
Recombinant DNA 287Ч288
Reconstruction 4Ч8 16
Reconstructionism 5Ч6
recycling 198 206 220 331 343 356
Red-blood-cell metabolism 408
Red-blood-cell model 400
Reddy, V.N. 64 483
Reder, C. 61 251 257 483
Reduced form 27
Reduced set of dependent variables 257 258
Reductionism 2Ч4 5Ч6
Reed, E. 361 475
refinement 298 327 366
Regan, L. 285 286 396 408 483
Regression, linear 148 149 150 173 176 186 191 414 419 451
Regression, nonlinear 172 173Ч174 175 176 178 189
Regular S-system 201Ч206 207
Regulation 12 18 39 57 62 86 160 179 208 223 261 329 400 401 405 407
Regulator 58 400 402
Regulatory mechanism 4 208 400 401
Regulatory pattern 7 58
Reich, J.G. 373 483
Reimers, J.M. 296 471 494
Reisig, W. 64 483
Relative changes 56 57 68 71 72 73Ч75 121 149 155 190 222 225 226 227 230 235 237 241 376 377 379 390 392 464
Relative concentrations 146 147 148 151
Relative growth 55Ч56
Relative process rate 146
Relnodes 64
Report interval 80 103 104 111
Repressible gene circuits 4
Repression 400
Research Collaboratory for Structural Bioinformatics (RCSB) 18
Residual 4 172 174
Resolution 107
Respiration 2 187 247 248 249
Response function 147
responsiveness 386 402
Results menu 44 106 110 111 112 114 117 130 250 444
Reverse engineering 63
Reverse reaction 32 91 162 188 317 372 462
Reverse transcriptase 17
Reversible/reversibility 32 96 155 163 164 168 170 190 298 300 334 349 447
Reversible/reversibility pathways 91Ч93 95
Reversible/reversibility reactions 25 26 91 92 162 167 272 304
Reversible/reversibility strategy 92
Rhee, S.G. 86 471
Ribeiro Filho, J.L. 174 483
Ribonuclease 346
Ribonucleotides 328 329 338
Ribose ring 12
Ribose-5-phosphate (R5P) 12 223 346 347 353
Riccati systems 60
Rigoulet, M. 323 483
Riley, M. 16 478 483
Risk assessment 139 411
Rivi, R. 17 482
RNA 56 327 330 346 347 348 351 352 353 363
RNase 348
Robustness 92 123 222 223 260 279 282 283Ч284 320 321 324 345 368 372 386 395 396 408
Robustness, glycolytic-glycogenolytic pathway 375Ч380 379Ч380
Robustness, tricarboxylic acid cycle model 306Ч315
Rodermel, S.R. 149 489
Rodnan, G.P. 185 479
Rodriguez, E 59 83 223 260 286 289 290 396 408 409 490
Rodriguez-Acosta, F. 490
Roessler 136 141
Rogers, J.R 18 470
Rolleston, F.S. 483
Roman, G.C. 38 483
Rose, I.A. 269 270 493
Rosen, O.M. 86 483
Rosenbloom, F.M. 334 359 476 487
Rothman, L. 273 483
Routh 210 453
Routh, E.J. 210 483
Rubin, C.S. 86 483
Rugen, P. 139 483
rule set 62
Run-time parameter 103
Rundles, R.W. 361 473
Russel, P.J. 263 489
Rust, P.F. 137 411 483 491
Rutherford, C.L. 294 295 493
S-adenosyl-L-methionine (SAM) 331 333 336 342 343 344 347 352
S-adenosylmethionine decarboxylase (SAMD) 334 348
S-AMP (adenylosuccinate) 330 331
S-distribution 164 411
S-phosphoribosyl-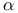 -l-pyrophosphate (PRPP) 12 328 334 335 349 351 362 363 461 -l-pyrophosphate (PRPP) 12 328 334 335 349 351 362 363 461
S-systems 59 60 129 174 191 193 209 210 217 219 251 288 307 404 405 410 411 452
S-systems in glycolytic-glycogenolytic pathway model 375
S-systems, 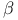 -term aggregation 277Ч278 -term aggregation 277Ч278
S-systems, allometric relationships in 55Ч56 70Ч73 74
S-systems, anaerobic fermentation pathway model 278Ч284
S-systems, analytical convenience 5 8
S-systems, bimolecular reactions 88Ч89
S-systems, branched pathways 81Ч82 83Ч84 112Ч113
S-systems, canonical models 65
S-systems, cascaded 87
S-systems, computer simulations applied to 97 99Ч103
S-systems, constraints 88Ч89
S-systems, degradation in 265 267 277 351 362 363 448 461
S-systems, dependent variables 77 79
S-systems, dynamics 54Ч55 282Ч283
S-systems, equation specification 70 74 76 77 78Ч80 82Ч84 92 94 99Ч103 277Ч278 338Ч340 375
S-systems, examples 52 54Ч55
S-systems, fluxes 83
S-systems, glucose-6-phosphate metabolism 98Ч99
S-systems, independent variables absent 201Ч204
S-systems, independent variables present 204Ч206
S-systems, irregular 206Ч208
| S-systems, irreversible representations 92
S-systems, kinetic orders 53Ч54 55 74 78 79 82
S-systems, linear pathway representation 71 78Ч80 81 115Ч119
S-systems, local stability 212
S-systems, log gains 280Ч282
S-systems, parameters 50 51Ч55 58 60 84
S-systems, phase-plane plots 110Ч111
S-systems, power functions 56Ч57 74
S-systems, precursor-product constraints 79Ч80 82 83 84
S-systems, properties of models 55Ч58 279Ч284
S-systems, purine metabolism 338Ч340
S-systems, rate constants 50 51Ч52 71 74 79 82
S-systems, regular 201Ч206
S-systems, reversible representations 92
S-systems, robustness of models 283Ч284
S-systems, sensitivities 282
S-systems, steady-state 119 123Ч124 201Ч208 280 407Ч409
S-systems, surprise pathway 115Ч119
S-systems, telescopic property 56Ч57 64
S-systems, theoretical justification 57Ч58 70Ч73
S-systems, two-variable 212
S-systems, validity 55Ч57 73
Saccharomyces cerevisiae 8 330 331
Saccharomyces cerevisiae, anaerobic fermentation pathway 260Ч292
Saccharopine 31 443
Sahota, A.S. 337 487
Salerno, C. 334 474
Salter, M. 149 488
Salvador, A. 223 405 483 484
Salvage pathway 129 328 329 334 335 341 349 351 354 462
Samitier, S. 184 488
Sampling design 139
Sanchez, J.-C. 9 493
Sands, P.J. 87 158 173 175 179 183 210 251 279 380 401 403 484 486 491
Sargent, R. 140 484
Sarrus's rule 208 211 213 432 433 442 468
Sato, K. 16 62 475
Saturation 51 57 146 160 163 186 270 273 369 374 378
Sauer, E 297 484
Sauro, H.M. 155 157 175 186 190 246 478
Savageau, M.A. 3 4 5 6 7 38 40 50 51 53 55 56 57 58 59 60 67 68 83 86 87 90 91 92 96 113 114 124 128 129 137 138 144 145 146 148 158 161 165 171 172 175 179 180 188 196 201 204 206 207 209 210 220 223 224 237 242 245 246 247 279 284 293 296 297 298 300 307 308 310 311 314 315 316 320 323 324 355 357 361 380 383 395 396 399 400 401 402 403 404 405 406 407 408 410 411 420 435 436 445 447 469 476 481 482 484 485 486 488 491
Savinell, J.M. 62 284 486
Scaffold 171
Scaling 161 267
Scenario 47 87 107 141 148 175 201 210 222 272 310 340 377 387 448
Schaeffer, H.J. 171 486
Schena, M. 17 149 486 492
Scholz, U. 15 62 477
Schomburg, D. 15 487
Schubauer-Berigan, M.K. 411 492
Schulze, E.-D. 149 489
Schurr, U. 149 489
Schuster, S. 38 60 61 119 223 245 246 251 284 476 478
Schwacke, L.H. 174 411 492 495
Scott, J.D. 171 473
Screening tool 8 290 327 362
Scrutton, M.C. 365 370 372 373 396 487
Search algorithm 353 361
Second-order approximation 406
Second-order reactions 53
Seegmiller, J.E. 334 359 476 487
Segel, I.H. 301 487
Segel, L.A. 38 487
Sel'kov, E.E. 373 483
Selectivity 402
Self-mutilation 327 328
Self-thinning 404
Selkov, E. 15 16 487
Selkov, E., Jr. 15 16 487
Semendjajew, K.A. 432 470
Semi-quantitative 359
Sensitivity 140 291 297 307 311 314 315 321 323 328 336 345 346 372 375 379 397 398 405 426 464
Sensitivity Analyses 222
Sensitivity analyses in GMA models 251Ч259
Sensitivity analyses of glycolytic-glycogenolytic pathway model 376Ч377 390Ч395
Sensitivity analyses with concentrations 235Ч241 252Ч253
Sensitivity analyses with fluxes 229Ч232 244Ч245 253Ч256
Sensitivity analyses with metabolites 224Ч229 233Ч243 254 255
Sensitivity analyses, connectivity relationships 246Ч249
Sensitivity analyses, constrained systems 239Ч241 257Ч258
Sensitivity analyses, examples 226Ч229 247Ч249 256Ч257
Sensitivity analyses, exercises 249Ч250 258Ч259
Sensitivity analyses, kinetic orders and 241Ч243 255Ч256 395
Sensitivity analyses, logarithmic gains and 224Ч233 252Ч254
Sensitivity analyses, purine metabolism model 342
Sensitivity analyses, ranking importance of processes 247Ч249
Sensitivity analyses, rate constants and 235Ч241 254Ч255 390Ч395
Sensitivity analyses, summation relationships 245Ч249
Sensitivity analyses, terminology 251Ч252
Sensitivity analyses, trajectories 250Ч251
Sensitivity analyses, transition time and 232Ч233
Sensitivity, aggregated 353
Sensitivity, anaerobic fermentation pathway model 282
Sensitivity, constrained 312 393 465
Sensitivity, deaggregated 363
Sensitivity, parameter 122Ч123 244Ч245
Sensitivity, profile 324 325 342 343 351 395 396 462
Sensitivity, types 223
Sensitivity, unconstrained 465
SEQUENCE 12 14 32 63 329
Sequence alignment 18
Seressiotis, A. 61 487
Serine 20Ч21 23 25 27
Serine phosphatase 25
Serum 40 208 357
Service, R.F. 6 487
Sethares, W.A. 174 494
Shalon, D. 17 486
Shannon, C.E. 5 487
Shimanouchi, T. 18 470
Shiraishi, E 38 113 129 145 161 165 171 172 188 209 223 246 284 293 296 297 298 308 310 311 314 315 316 320 323 324 407 475 487
Shonkwiler, R.W. 131 494
Shott, S. 140 487
Sicilia, J. 246 480
Signals, biochemical 9 14 17 47 53 63 86 93 136 187 248 284 314 320 330Ч331 397 443 444
Siljak, D.D. 222 487
Simmonds, H.A. 337 355 470 487
Simon, W. 295 494
Simonds, H.A. 337 364 490
simplification 5 6Ч7 37 57 262 325 330 366 373 456
Simpson, G.G. 2 4 488
Simulation 41 157 158 179 230 237 243 297 324 325 345 356 357 360 361 363 368 378 387 392 403 see
Simulation Monte Carlo 138Ч140
Sine waves 110 111 125 141 449
Single-displacement mechanism 269
Skupp, S. 343 345 469
Slein, M.W. 374 488
Slopes 42 67 68 74 147Ч149 151 154 174Ч184 233 234 235 270 278 291 377 417 419 420 423 449
Slug 293 315
Small, J.R. 62 149 156 284 473 488
Smith, E.L. 365 493
Smith, G.K. 149 488
Smith, S. 63 493
Smoothing algorithm 175
Sniegoski, C.A. 62 63 488
Solution 158 437
Solution algebraic 190 383
Solution analytical 65 66 75 210 350 353 406 410
Solution dynamical 84 85 119 120 122 124 126 280 353 361 448 456
Solution numerical 44 65Ч68 75 85 108 119 123 132Ч133 143 174 195 205 207 216 219 315 357 389 406 435
Solution, parameter 104 107 108
Solution, points 45 444
Solution, space 207
Somero, G.N. 160 477
Somogyi, R. 62 63 480 488 489 493
Sonati, E 17 482
Sorocarp 293 296
Sorribas, A. 59 60 83 91 92 96 98 113 144 145 146 148 149 150 164 171 172 184 185 223 242 246 260 261 268 270 272 273 275 279 281 283 284 300 314 326 327 328 335 336 338 341 352 355 357 360 361 383 395 399 405 407 411 435 447 471 472 476 483 486 488 492
Spatial organization 7
Spearman's rank correlation coefficient 140
|
|
 |
| –еклама |
 |
|
|
 |
|
ќ проекте
|
|
ќ проекте



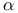 -l-pyrophosphate (PRPP)
-l-pyrophosphate (PRPP) 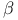 -term aggregation
-term aggregation